iPHACTORY
iPHACTORY ist ein gemeinsames DFG-Projekt mit Anna Matuszyńska (Heinrich-Heine-Universität Düsseldorf), die das Teilprojekt Metabolic Modeling bearbeitet. Mit einer Kombination aus Transcriptomics- und Metabolomics-Ansätzen konnten wir in früheren Arbeiten zeigen, dass glanduläre Trichome der Tomate, obgleich sie photosynthetisch aktiv sind, Kohlenstoff in Form von Saccharose importieren (Balcke et al., 2017). Demnach dient die Photosynthese in den Trichomen der Energiegewinnung und Bereitstellung von Reduktionsäquivalenten für die Biosynthese von Terpenoiden. Darüber hinaus vermuten wir, dass erzeugtes CO2 durch die Ribulose-1,5-Bisphosphatcarboxylase (RubisCo) wieder recycelt wird. Dies geschieht entweder als Teil eines vollständigen Calvin-Bassham-Benson- (CBB)-Zyklusses oder auf einem Umweg über den Pentosephosphatweg. Um diese Hypthese zu überprüfen, entwickeln wir verschiedene Tomatenlinien, bei denen bestimmte Gene entweder inaktiviert, herunterreguliert oder überaktiviert werden. Das betrifft die Gene für eine trichomspezifische Isoform der kleinen Untereinheit von RubisCo, für die Phosphoenolpyruvat-Carboxykinase (PEPCK) und für die ATP-Citratlyase. Metabolomics-Analysen der Mutanten sollen anschließend die nötigen Daten liefern, um unser Modell schrittweise zu präzisieren.
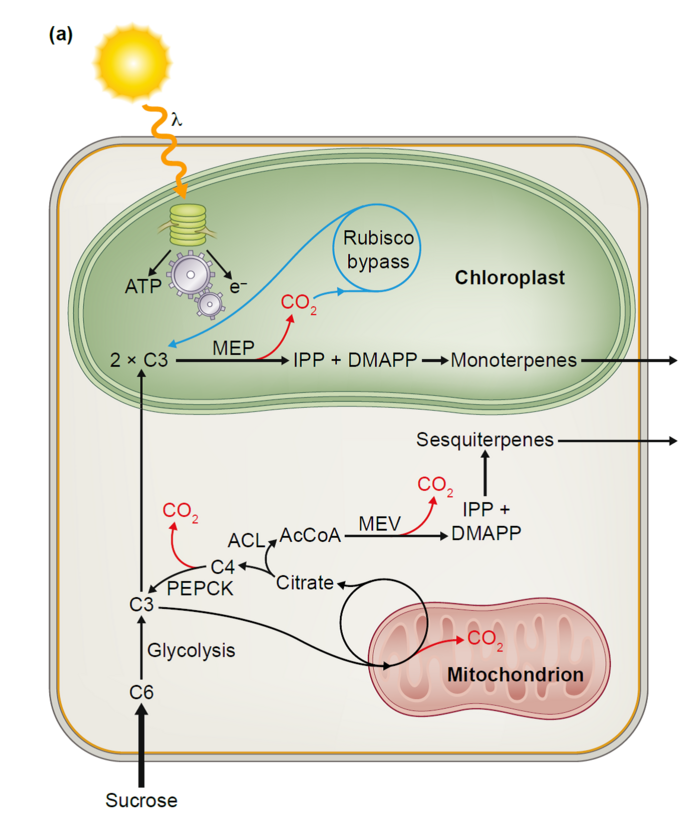
Vereinfachtes Modell von photosynthetisch aktiven glandulären Trichomen (von Schuurink & Tissier, 2019). Die Hauptprodukte sind Monoterpene und Sesquiterpene, die aus dem plastidiären Methyl-Erythritol-Phosphat-Weg (MEP) bzw. dem Mevalonat-Weg (MEV) stammen. Obgleich die Trichome photosynthetisch aktiv sind, importieren sie Saccharose aus dem Blattgewebe, die über die Glykolyse zu C3-Produkten umgewandelt wird. Die Bereitstellung von Acetyl-CoA (AcCoA) im Zytosol wird durch den Citrat-Shuttle mit den Mitochondrien gesichert, da AcCoA die Mitochondrienmembran nicht passieren kann. Der erste Reaktionsschritt des Citratcyklus‘ in den Mitochondrien produziert Citrat, das ins Zytosol exportiert wird, wo es durch die ATP-Citratlyase (ACL) in Oxalat (C4) und AcCoA aufgespalten wird. Die so produzierten C4-Dicarbonsäuren können dann entweder durch die Phosphoenolpyruvat-Carboxykinase (PEPCK) wieder in eine C3-Verbindung (in diesem Fall Phosphoenolpyruvat) umgewandelt oder in die Mitochondrien zurückgeführt werden. Auch C3-Triosephosphate können in die Chloroplasten transportiert werden. Hier dienen sie als Vorläufer für den plastidiären MEP-Weg. Im Chloroplasten liefert die Lichtreaktion der Photosynthese die benötigte chemische Energie (ATP) und auch die Reduktionsäquivalente. Das in großen Mengen freigesetzte CO2 (rot markiert) wird vermutlich durch die Ribulose-Bisphosphat-Carboxylase (RubisCo) im sogenannten RubisCo-Bypass refixiert.
Am Projekt beteiligt sind:
Diese Seite wurde zuletzt am 07 Nov 2019 11 Dec 2024 12 Nov 2019 geändert.

