Natürliche Variation von Auxin-abhängiger pflanzlicher Entwicklung
Mit über 1000 öffentlich verfügbaren natürlichen Akzessionen von Arabidopsis thaliana, darunter viele mit komplett sequenziertem Genom, stellt die natürlich vorkommende genetische Variation im Arabidopsis thaliana Genpool eine wetvolle Ressource für die genetische Analyse wichtiger Merkmale dar.
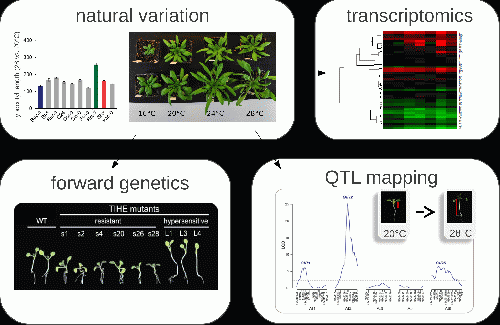
Wir nutzen diese natürliche Ressource, um in diesem Zusammenhang Entwicklungs- und Adaptionsprozesse besser zu verstehen, die vom Phytohormon Auxin reguliert werden. In einem Screening von mehreren hundert natürlichen Akzessionen konnten wir eine große Spanne an natürlicher Variation für auxinabhängige Phänotypen identifizieren (Delker et al., 2008). Unser Hauptinteresse gilt nun der Aufklärung der zugrundeliegenden genetischen Zusammenhänge, was wir in erster Linie mit quantitativ genetischen (QTL, GWAS) und transkriptionellen Ansätzen verfolgen (Delker et al., 2010; Delker und Quint, 2011). Mit einem komplementären Ansatz analysieren wir die Selektionsmuster der Gene, die die verschiedenen Level des Auxinweges regulieren, mit populationsgenetischen Ansätzen.
Temperatur Signaling
Die Umgebungstemperatur ist einer der wichtigsten abiotischen Faktoren, der nahezu alle Aspekte pflanzlicher Morphologie und Entwicklung beeinflusst. Die Reaktion auf extremen Temperaturstress (Frost/Hitze) wurde in der Vergangenheit ausgiebig untersucht. Pflanzen zeigen jedoch bereits bei geringen Temperaturerhöhungen z.T. dramatische morphologische und entwicklungsspezifische Veränderungen. Die molekularen Grundlagen dieser Reaktion sind aber bislang nur wenig untersucht und kaum verstanden. Wir nutzen die natürliche Variation im weltweiten Arabidopsis thaliana Genpool und verfolgen quantitative und klassische (forward) genetische Ansätze sowie Transkriptomanalysen. Das Ziel ist die Identifizierung molekularer Komponenten und regulatorischer Hierarchien der planzlichen Reaktionen auf moderat erhöhte Umgebungstemperaturen.
Evo-Devo
Es werden verschiedenste Ansätze verwendet, um die Evolution pflanzlicher Entwicklung zu untersuchen. Ein wichtiges Tool in diesem Zusammenhang sind phylogenetische Ansätze. Wir nutzen Phylogenetik, um zwei verschiedene Bereiche zu analysieren:
(i) Zum einen rekonstruieren wir die evolutionäre Geschichte/Vergangenheit von Genfamilien, die eine wichtige Rolle in der pflanzlichen Entwicklung spielen (Schumann et al., 2011; Janitza et al., 2012). Darüber hinaus
(ii) kombinieren wir Phylogenetik mit Transkriptomanalysen (Phylotranscriptomics). Wir verwenden Phylotranscriptomics, um klassische Entwicklungsprozesse wie Embryogenese in Pflanzen zu analysieren (Quint et al., 2012). Im Mittelpunkt unseres Interesses steht dabei das sogenannte Developmental Hourglass Konzept.
Diese Seite wurde zuletzt am 06 Jun 2012 26 Apr 2013 26 Apr 2013 26 Apr 2013 geändert.