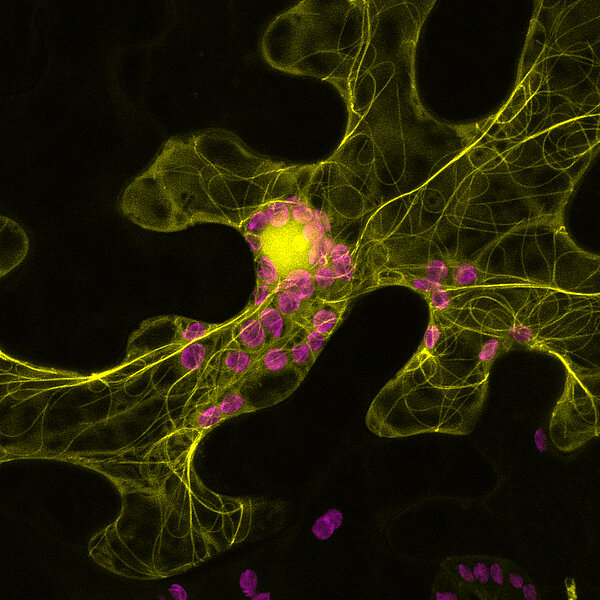
Xanthomonas-Effektorprotein bewirkt Abbau von pflanzlichen Mikrotubuli.
Xanthomonaden verursachen als pflanzenpathogene Bakterien enorme Schäden im Kultur- und Zierpflanzenanbau. Sie befallen mehr als 400 Pflanzenarten, darunter Weizen und Reis, Weißkohl, Walnüsse, Zitronen, Baumwolle und Sojabohnen, aber auch Begonien, Pelargonien und Hyazinthen. Im Laufe der Evolution haben die Erreger zahlreiche Strategien entwickelt, wie sie die unterschiedlichen Pflanzenarten erfolgreich infizieren und besiedeln können. Wissenschaftler/innen des IPB und der MLU haben jüngst eine dieser Infektionsstrategien näher beleuchtet und neue Erkenntnisse zu diesem speziellen Mechanismus der Krankheitsentstehung zusammengetragen.
Ganz generell verschaffen sich Xanthomonas-Bakterien durch Wunden oder durch die Spaltöffnungen Zugang zum Pflanzengewebe und vermehren sich, je nach Erregerart, entweder in den Leitbahnen oder im Interzellularraum zwischen den Mesophyllzellen der Blätter. Über nadelartige Strukturen des Typ-III-Sekretionssystems injizieren die Bakterien zudem Typ-III-Effektorproteine (T3Es) direkt ins Zytosol der Pflanzenzellen. Hier unterdrücken die Effektorproteine die basale Immunreaktion der Pflanzen. Das führt bei empfindlichen Pflanzen zu nekrotischen Flecken von abgestorbenem Gewebe und insgesamt zu Welke und Fäule von Blättern und Stängeln.
Jeder Bakterienstamm oder Pathovar hat dabei in Interaktion mit seiner Wirtspflanzenart sein ganz eigenes Arsenal an verschiedenen Effektorproteinen entwickelt. So kennt man beispielsweise für den Erreger der Bakterienfleckenkrankheit von Tomaten und Paprika Xanthomonas campestris pathovar vesicatoria (auch bekannt als X. euvesicatoria) 36 verschiedene Effektorproteine, während im Kohlpathogen Xanthomonas campestris pv. campestris bisher 24 spezielle T3Es entdeckt hat. Die Aufklärung der vielfältigen biochemischen Aktivitäten der T3Es innerhalb der Pflanzenzellen steht seit 25 Jahren im Fokus der Pflanzenforschung. Manche Effektorproteine können die Transkription in der Wirtszelle aktivieren, andere sind in der Lage, Wirtsproteine zu phosphorylieren oder zu ubiquitinieren und damit dem Abbau freizugeben; von vielen der bisher gefundenen T3Es weiß man jedoch bisher noch nicht, was sie bewirken und welchen pflanzlichen Signalweg sie stören.
Trotz ihrer Vielfalt in Struktur und Wirkung von Pathovar zu Pathovar gibt es einige T3Es mit konservierten strukturellen Merkmalen, die in den meisten sequenzierten Xanthomonas-Stämmen vorhanden sind. Dazu gehört das Xanthomonas Outerprotein L (XopL), das als angestammter Effektor zur Virulenz mehrerer Xanthomonas-Stämme beiträgt, die verschiedene Pflanzenarten infizieren. XopLs weisen eine konservierte leucinreiche Region und eine E3-Ligase auf, ein Enzym, das Ubiquitin auf andere Proteine überträgt und sie damit zum Abbau markiert. Trotz dieser konservierten Eigenschaften gibt es neue Hinweise von den Hallenser Wissenschaftlern, die darauf hindeuten, dass es funktionelle Unterschiede zwischen den XopLs verschiedener Xanthomonas-Arten gibt. Die Expression von XopLs in Tabakpflanzen ergab zwei wichtige Erkenntnisse: XopL-Proteine verschiedener Xanthomonas-Arten unterscheiden sich sowohl in ihrer Fähigkeit, den pflanzlichen Zelltod auszulösen, als auch in ihrer Lokalisation innerhalb der Pflanzenzelle. Als ein mögliches Zielprotein für die XopL-Ligase für drei der vier getesteten Xanthomonas-Arten konnten die Hallenser Pflanzenexperten die Mikrotubuli des pflanzlichen Zytoskeletts identifizieren.
Durch vergleichende Sequenzanalysen der verschiedenen XopL-Proteine konnten die Forscher schließlich eine prolinreiche und eine α-helikale Region als jene Strukturen identifizieren, die für die Mikrotubuli-Bindung der E3-Ligasen verantwortlich ist und einen Zusammenhang zwischen Mikrotubuli-Bindung und Zelltod-Phänotypen herstellen. Wie genau diese XopL-Mikrotubuli-Interaktion zur Einleitung des pflanzlichen Zelltods führen, ist noch nicht klar. Mikrotubulis spielen vermutlich eine Rolle bei der Signalübertragung pflanzlicher Immunreaktionen und auch bei bestimmten Abbau- und Entsorgungsprozessen der Autophagie. Mit ihrer Studie zeigten die Wissenschaftler beispielhaft, wie die Untersuchung eines Effektorproteins im Kontext einer Gattung und nicht einer einzelnen Spezies wichtige Erkenntnisse über seine Lokalisierung und Aktivität liefern kann.