Verbundprojekt TP1:
In-Silico-Screening nach neuen Wirkstoffen zur Erhöhung der Stickstoffeffizienz
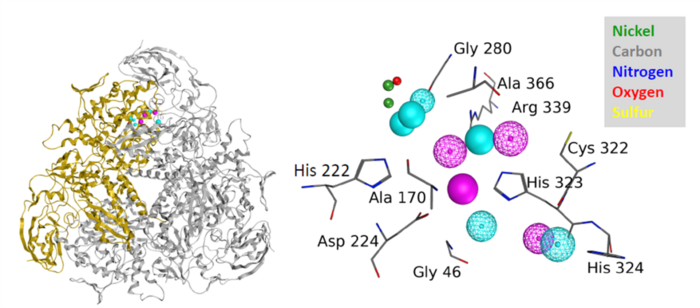
Ziel dieses Teilprojektes (für alle Projekte s. http://www.aip.uni-halle.de/projekte) ist es, Strukturen für Urease- und Nitrifikationsinhibitoren zu identifizieren oder gegebenenfalls neu zu entwerfen und nach ersten Biotests zu optimieren. Die Inhibitoren sollen nicht nur hochwirksam sein, sondern auch günstige Eigenschaften hinsichtlich Anwendung im Feld, Stabilität, Toxikologie und Abbaubarkeit aufweisen. Ein weiteres Ziel war die Suche nach adressierbaren (drugable) Targets im N-Stoffwechsel der Pflanzen, um deren Stickstoff-Effizienz zu verbessern. Konkret wurde ein Modell der Urease-Bindungsstelle erstellt, ein geeignetes Pharmakophor generiert (Abb. 1) und eine umfassende Suche nach geeigneten Liganden in MOE und in hauseigenen Datenbanken durchgeführt. Ein alternativer Ansatz stützte sich auf einen Protein-Protein-Interaktionsinhibitor (PPII). Zu diesem Zweck wurden makrozyklische Peptoide synthetisiert und in einem Bodenmodellversuch getestet. Im Vergleich zu kommerziellen Ureaseinhibitoren wurde jedoch keine Wirkung gefunden. Darüber hinaus wurde ein Homologiemodell für ein ammoniumverarbeitendes Enzym entwickelt. Auf der Grundlage dieses Modells wurden durch virtuelles Screening 42 potenzielle Inhibitoren identifiziert. In einem Bodentest zeigte eine der Substanzen eine ausgeprägte Hemmung der Nitrifikation.
Diese Seite wurde zuletzt am 19 May 2020 19 May 2020 geändert.