|
Leibniz-Institut für Pflanzenbiochemie
|
|
| Durchbruch bei CRISPR/Cas: Optimierte Genschere erlaubt den stabilen Einbau von großen Genen |
|
| Großer Fortschritt an der CRISPR-Front. Wissenschaftlern des Leibniz-Instituts für Pflanzenbiochemie (IPB) ist es gelungen, sehr effizient große Gen-Abschnitte stabil und präzise in die DNA von höheren Pflanzen einzubauen. Dafür optimierten sie die Gen-Editierungsmethode CRISPR/Cas, die gemeinhin als Genschere bekannt ist. Das verbesserte CRISPR-Verfahren bietet sowohl für die Züchtung als auch für die Wissenschaft großartige Möglichkeiten der gezielten Veränderung des Erbguts von höheren Pflanzen. Die Studie wurde von Prof. Alain Tissier und Dr. Tom Schreiber geleitet und ist im renommierten Fachjournal Molecular Plant erschienen. |
|
|
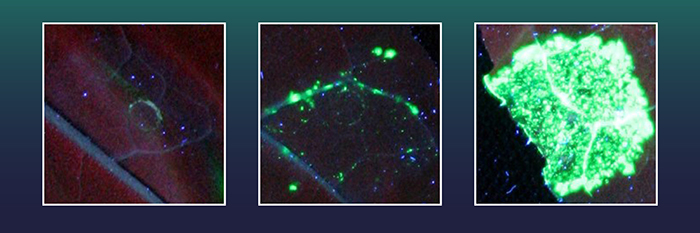 |
| Lokale Änderung des Erbguts mit Hilfe des Gen-Editierungs-Verfahrens CRISPR/Cas in den Blättern des australischen Tabaks Nicotiana benthamiana. Jeder grün fluoreszierende Punkt steht für eine Blattzelle, in der erfolgreich ein großer Genabschnitt ins Erbgut der Pflanze eingefügt wurde. Für diese Versuchsreihe wurden verschiedene Varianten der Genschere Cas9 eingesetzt; einmal eine inaktive Variante von Cas9 (linkes Bild), eine aktive Cas9-Variante (Mitte) und eine Cas9-Variante, die mit einem weiteren Enzym, einer Exonuklease gekoppelt ist (rechts). Erst der Einsatz der Exonuklease bewirkt eine starke Zunahme der korrekten Gen-Einbau-Ereignisse. |
|
CRISPR/Cas ist ein Verfahren mit enormem Potential für die gezielte Veränderung von einzelnen Genen. Das gilt jedoch längst noch nicht für alle genetischen Umgestaltungen, die auf der Wunschliste von Züchtern und Wissenschaftlern stehen. Für ein Knock-Out von Genen, also das Ausschalten oder Entfernen von bereits vorhandenen Genen, eignet sich die Genschere hervorragend, nicht aber für ein punktgenaues Einfügen von Genen oder den Austausch von Genabschnitten. Für dieses gezielte Knock-In von Genen in das Erbgut von höheren Pflanzen ist die Genschere bisher zu ineffizient und deshalb nur bedingt geeignet.
„Der Grund dafür liegt in der pflanzeninternen Reparaturmaschine für DNA-Brüche“, sagt Dr. Tom Schreiber. Diese Reparaturenzyme sind sofort zur Stelle, sobald Schäden an der DNA auftreten. Sie erkennen auch die glatten Schnitte, die von der Genschere gesetzt werden, und fügen die beiden durch- trennten DNA-Stränge der Doppelhelix sofort wieder zusammen. Dieses Kitten der zerschnittenen DNA erfolgt sehr schnell und nicht sehr genau; es kommt zu kleineren Informationsverlusten, bei dem winzige DNA-Abschnitte verloren gehen oder auch hinzugefügt werden. „Diese Ungenauigkeiten sind bei Knock-Out-Vorhaben nicht schlimm und sogar erwünscht“, sagt Tom Schreiber, „denn ich will ja das Gen sowieso ausschalten. Aber wenn ich ein Gen einfügen will, dann muss das sehr präzise geschehen. Die genetische Information muss exakt eingesetzt werden, es darf nicht ein Baustein fehlen und es darf auch kein einziger Baustein zusätzlich mit integriert werden, sonst verliert das Gen seine Funktion und der gesamte Versuch war umsonst.“
Aus diesem Grund ist ein solcher präziser und narbenfreier Einbau von größeren Genen oder DNA-Abschnitten mit CRISPR/Cas bisher nur in seltenen Einzelfällen geglückt. Um die Erfolgsquote beim Gen-Einbau zu erhöhen, haben die Hallenser Wissenschaftler die Genschere mit einem weiteren Enzym, einer sogenannten Exonuklease, versehen. Exonukleasen können die von der Genschere gesetzten DNA-Schnittstellen derart verändern, dass die zellinternen Reparaturenzyme den DNA-Schaden nicht mehr erkennen und ihn folglich nicht wieder zusammenkleben können. Der durch CRISPR/Cas einzubauende DNA-Abschnitt hätte demnach genügend Zeit, um sich - mit Hilfe eines weiteren und sehr präzisen zellulären Reparaturmechanismus - an der richtigen Stelle im Erbgut zu integrieren.
Im Experiment haben die Hallenser Wissenschaftler verschiedene Exonukleasen viralen, bakteriellen, pflanzlichen und menschlichen Ursprungs auf ihre Fähigkeit getestet, die Anzahl der präzisen Gen-Einbau-Ereignisse zu erhöhen. Dafür brachten sie die Genschere mit den entsprechenden Exonukleasen und mit dem einzubauenden Gen X-Abschnitt in die Blattzellen der Tabakpflanze Nicotiana benthamiana ein. Diese Tabakzellen waren zuvor von den Forschern mit einem Gen für einen grün fluoreszierenden Farbstoff ausgestattet worden. Und sie enthielten außerdem ein zerstörtes Gen X, das für die Bildung dieses grün fluoreszierenden Farbstoffes benötigt wird. Die Bildung des Fluoreszenzfarbstoffes konnte aber nicht stattfinden, weil ein großer Teil der genetischen Information von Gen X fehlte.
Nur wenn der fehlende Genabschnitt von X mittels CRISPR/Cas präzise wieder eingefügt und das Gen X damit wieder repariert wird, kann sich der grüne Farbstoff bilden. Alle Zellen mit geglücktem Gen-Einbau fluoreszieren dann grün und die Quote des erfolgreichen Gen-Einbaus kann somit gezählt werden. Zwei der getesteten Exonukleasen, darunter eine aus der Familie der Herpesviren, erwiesen sich dabei als besonders wirksam. Mit ihrer Hilfe erreichte das Hallenser Team 38-mal mehr perfekte Gen-Einbau-Ereignisse, als mit CRISPR/Cas allein.
Dieser Versuchsansatz wurde anschließend mit anderen einzubauenden Genen und an anderen Pflanzen, nämlich an der Ackerschmalwand und an Weizen, getestet. Da der Gen-Einbau in den Tabakpflanzen nur lokal in den Blättern stattfand, ging das integrierte Gen in der nächsten Tochtergeneration wieder verloren, war also nur zeitlich begrenzt im Erbgut vorhanden. Bei der Ackerschmalwand und beim Weizen haben die Hallenser CRISPR-Experten deshalb versucht, das zu integrierende Gen so ins Erbgut der Keimbahn einzubauen, dass es stabil an die kommenden Pflanzengenerationen weitervererbt wird. Auch der stabile, also vererbbare Knock-In der Gene gelang mit Hilfe der getesteten Exonukleasen bei der Ackerschmalwand mit einer zehnfach erhöhten Häufigkeit und beim Weizen bei über einem Prozent der Tochterpflanzen. „Ein Prozent klingt jetzt erst mal nicht viel“, führt Tom Schreiber aus, „aber wenn ein Züchter eine bestimmte Eigenschaft in seine Pflanze bringen will, dann müsste er mit unserem optimierten CRISPR/ Cas-Verfahren nur etwa 50-100 Tochterpflanzen der ersten Generation durchforsten, um eine Pflanze mit dem gewünschten Merkmal zu finden. Das wäre eine erhebliche Zeitersparnis gegenüber herkömmlichen Züchtungsmethoden, wo man zu diesem Zweck 500 bis 1000 Pflanzen analysieren müsste.“
Fazit: Die von den Hallenser Wissenschaftlern optimierte CRISPR/Cas-Methode ist ein vielversprechendes Werkzeug für den gezielten Einbau von Genen in höhere Pflanzen und möglicherweise auch in andere Organismen. Künftig könnten Pflanzenzüchter mit diesem Verfahren beispielsweise verlorengegangene Resistenzgene gegen Krankheitserreger aus Wildarten oder alten Kultursorten in die modernen, ertragreichen Elitesorten wieder einfügen. Das und viele weitere wünschenswerte Eigenschaften könnten auf diese Art ihren Einzug in die Pflanzenzüchtung halten und zur Entwicklung von robusteren Kultursorten beitragen. Für die Wissenschaft bietet dieser Ansatz grandiose Möglichkeiten, bestimmte pflanzeneigene Gene elegant und in einem Schritt durch modifizierte Kopien ihrer selbst auszutauschen. Das ist besonders hilfreich bei der Funktionsaufklärung dieser Gene. |
|
|
|
Originalpublikation:
Tom Schreiber, Anja Prange, Petra Schäfer, Thomas Iwen, Ramona Grützner, Sylvestre Marillonnet, Aurélie Lepage, Marie Javelle, Wyatt Paul & Alain Tissier. Efficient scar-free knock-ins of several kilobases in plants by engineered CRISPR/Cas endonucleases. Molecular Plant 2024, doi: 10.1016/j.molp.2024.03.013.
Weitere fachliche Informationen:
Sylvia Pieplow, Neue CRISPR-Methode erlaubt stabiles Knock-In von großen Genen. IPB-Webseite, Newsticker Wissenschaft |
|
Herausgeber: Leibniz-Institut für Pflanzenbiochemie | Pressestelle | Sylvia Pieplow
Weinberg 3 | 06120 Halle (Saale) | E-Mail: spieplow@ipb-halle.de | Tel: +49 (0) 345 5582 1110
|
|
|